sábado, 30 de noviembre de 2013
Diseccionando los efectos del ganado sobre la vegetación
jueves, 23 de mayo de 2013
Dos recomendaciones: Divúlgame y Edublog
Hace ya unos tres años que compañeros y amigos del Departamento de Ecología de la Universidad Autónoma de Madrid, donde realicé mi tesis doctoral, mantienen un blog que nació en el ámbito del Máster Interuniversitario de Ecología que imparten junto a la Complutense: Edublog Ecología. A pesar de que en principio los contenidos van orientados a los alumnos de este máster, también podemos encontrar contribuciones muy interesantes de divulgación en el ámbito de la Ecología. Un ejemplo de ello es el reciente post de Francisco Martín Azcárate, profesor e investigador del departamento, sobre la existencia o no de disenso en la comunidad científica sobre las causas del cambio climático. En definitiva, un blog que debería estar en vuestros feeds para estar enterados de lo que se hace en la actualidad en una de las instituciones universitarias más dinámicas en investigación de ecología de pastos, aves esteparias, socioecosistemas y mucho más.
La segunda recomendación es la de Divúlgame.org, que hace dos días que acaba de nacer de su hermana Divúlgame.net. Casi todo el mérito de estos dos portales se debe a su principal perpetrador, Adrián Muñoz, estudiante de Ingeniería de Sistemas de Telecomunicaciones. Ya seguía el "Menéame" de ciencia, Divúlgame.net, y desde hace ya casi un año, Adrián planteó el crear una web con todo el que quisiera cuyos principales contenidos fueran traducciones al español de textos académicos. Yo me propuse en principio a colaborar, pero diversas obligaciones y responsabilidades me lo han impedido (lo siento, Adrián). Aún falta gente que quiera colaborar, así que no dudéis en poneros en contacto con la administración del sitio o inscribiros en su grupo de Google Groups.
Les deseo larga vida a estos dos proyectos. Enhorabuena y gracias a los que los hacen posibles.
miércoles, 22 de mayo de 2013
Celestia y el próximo sobrevuelo de Cassini sobre Titán
domingo, 24 de marzo de 2013
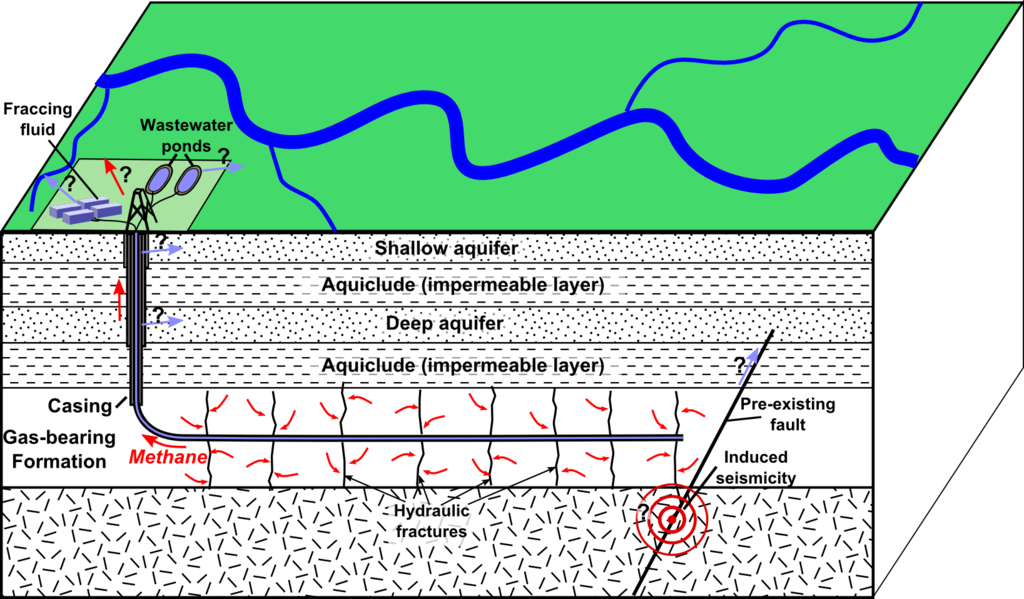
Fracking: fracturando el suelo, ¿o nuestro futuro?
"El debate sobre el fracking llegó al gran público en 2011 cuando el entonces lehendakari vasco, Patxi López (PSE), anunció por todo lo alto que en Euskadi se había encontrado una reserva de gas pizarra equivalente a cinco veces el consumo anual de toda España. Su Ejecutivo respaldó el proyecto, llamado Gran Enara, a través de la sociedad pública Hidrocarburos de Euskadi. El Gobierno de Iñigo Urkullu (PNV) le puso freno nada más ganar las elecciones"
martes, 12 de febrero de 2013
Feliz Día de Darwin
La labor de Darwin nos dio como resultado nuestro justo lugar en este Universo. Ya no estamos arrinconados al margen del resto de seres vivos (al menos aquí, en la Tierra), sino que compartimos una herencia ancestral con todos ellos, somos parte de un noble linaje de cientos de millones de años. Eso ha supuesto que nos dignifiquemos aún más si cabe al hacernos más humildes y más respetuosos con las especies de las que somos primos lejanos con las que convivimos en esta pequeña roca perdida en el brazo de una galaxia. Aún más, los trabajos de Darwin representaron el primer paso para empezar a desterrar definitivamente la idea de que hay diferencias entre nosotros que justifiquen grotescas diferencias de trato: todos somos miembros de una única especie descendiente de un ancestro común.
Para celebrar el nacimiento de Darwin quiero regalaros una cita de El origen de las especies sobre uno de los motores de la evolución, descubierto simultáneamente por Darwin y Wallace, cuya elegancia y sencillez lleva en no pocas ocasiones a equívocos al intentar explicar los hechos complejos a los que hace referencia: la Selección Natural.
¡Feliz Día de Darwin!
Metafóricamente puede decirse que la selección natural está buscando cada día y cada hora por todo el mundo las más ligeras variaciones; rechazando las que son malas; conservando y sumando todas las que son buenas; trabajando silenciosa e insensiblemente, cuando quiera y dondequiera que se ofrece la oportunidad, por el perfeccionamiento de cada ser orgánico en relación con sus condiciones orgánicas e inorgánicas de vida. Nada vemos de estos cambios lentos y progresivos hasta que la mano del tiempo ha marcado el transcurso de las edades; y entonces, tan imperfecta es nuestra visión de las remotas edades geológicas, que vemos sólo que las formas orgánicas son ahora diferentes de lo que fueron en otro tiempo.
Darwin, Ch. R. 1859. El origen de las especies.
martes, 27 de noviembre de 2012
R: Curvas de Respuesta Principal (PRC) 3. Significatividad de los ejes
Se nos quedó en el tintero en nuestros análisis PRC un tema algo ecabroso y complicado que es el análisis de la significatividad de los ejes. El test de Monte Carlo para el primer eje no tiene problema en vegan con la función anova.cca:
## Significación primer eje
anova(mod, strata = week, perm.max = 499, first = T)
## Permutation test for rda under reduced model
## Permutations stratified within 'week'
##
## Model: prc(response = pyrifos, treatment = dose, time = week)
## Df Var F N.Perm Pr(>F)
## RDA1 1 25.3 15.1 199 0.005 **
## Residual 77 129.0
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
En la última versión de vegan (2.0-5) se introdujo la modificación adecuada en esta función para que se pudiera utilizar el parámetro by con la opción “axis” con un objeto proveniente de de la función prc. Supuestamente con este parámetro la función realiza los test de Monte Carlo para todos los ejes extrayendo los scores de cada eje y utilizándolos como covariables para el siguiente en cada paso, tal como debe hacerse para garantizar la independencia de cada uno de ellos:
anova(mod, strata = week, perm.max = 499, by = "axis")
## Model: rda(formula = pyrifos ~ dose * week + Condition(week))
## Df Var F N.Perm Pr(>F)
## RDA1 1 25.3 15.10 199 0.005 **
## RDA2 1 8.3 4.95 199 0.005 **
## RDA3 1 6.0 3.61 199 0.005 **
## RDA4 1 4.8 2.85 199 0.005 **
## RDA5 1 4.1 2.48 199 0.005 **
## RDA6 1 3.9 2.30 199 0.005 **
## RDA7 1 3.6 2.14 199 0.010 **
## RDA8 1 3.3 1.99 199 0.005 **
## RDA9 1 3.1 1.84 299 0.017 *
## RDA10 1 2.6 1.52 499 0.048 *
## RDA11 1 2.5 1.47 499 0.066 .
## RDA12 1 2.2 1.32 199 0.115
## RDA13 1 2.1 1.27 99 0.130
## RDA14 1 1.9 1.16 99 0.340
## RDA15 1 1.8 1.07 99 0.430
## RDA16 1 1.6 0.97 99 0.570
## RDA17 1 1.6 0.94 99 0.590
## RDA18 1 1.4 0.86 99 0.730
## RDA19 1 1.4 0.83 99 0.700
## RDA20 1 1.3 0.77 99 0.810
## RDA21 1 1.2 0.72 99 0.870
## RDA22 1 1.1 0.68 99 0.940
## RDA23 1 1.0 0.60 99 0.980
## RDA24 1 0.9 0.55 99 1.000
## RDA25 1 0.9 0.51 99 1.000
## RDA26 1 0.8 0.47 99 0.980
## RDA27 1 0.7 0.45 99 1.000
## RDA28 1 0.7 0.43 99 1.000
## RDA29 1 0.7 0.41 99 1.000
## RDA30 1 0.6 0.36 99 1.000
## RDA31 1 0.6 0.35 99 1.000
## RDA32 1 0.5 0.32 99 1.000
## RDA33 1 0.5 0.31 99 1.000
## RDA34 1 0.4 0.26 99 1.000
## RDA35 1 0.4 0.25 99 1.000
## RDA36 1 0.4 0.24 99 1.000
## RDA37 1 0.4 0.22 99 1.000
## RDA38 1 0.3 0.20 99 1.000
## RDA39 1 0.3 0.20 99 1.000
## RDA40 1 0.3 0.18 99 1.000
## RDA41 1 0.3 0.17 99 1.000
## RDA42 1 0.3 0.16 99 1.000
## RDA43 1 0.2 0.12 99 1.000
## RDA44 1 0.2 0.11 99 1.000
## Residual 77 129.0
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Sin embargo, ya adelanto que los resultados de estos análisis no son correctos. Esto lo he comprobado con resultados obtenidos con CANOCO de otra serie de datos propios. El caso es que la función en vegan parece no tomar las puntuaciones adecuadas de cada eje. He dejado esta cuestión en la página de del foro de vegan por si se tratara de un bug.
A la espera de conocer qué está pasando realmente con el parámetro by de la función anova.cca, tenemos una solución alternativa para comprobar la significatividad de los ejes de nuestra PRC subsiguientes al primero. Lo primero es saber que una PRC no es más que una RDA de la forma rda (species ~ Treatment * Year + Condition (Year)). Lo segundo que necesitamos saber es dónde están las puntuaciones de los puntos de muestreo que son combinaciones lineales de las variables ambientales y que son las que tenemos que utilizar como covariables en el análisis del siguiente eje (las SamE de CANOCO). El resultado de la PRC es un objeto cca.object y dichas puntuaciones se encuentran en cca.object$CCA$u. Así que solo tenemos que extraerlas y realizar un RDA añadiéndolas como covariable (argumento Condition). Por último, realizaremos el test de Monte Carlo para el primer eje de este análisis que correspondería a nuestro segundo eje de PRC.
## Significación segundo eje
SamE <- mod$CCA$u
SamE <- SamE[, 1]
secondaxis <- rda(pyrifos ~ dose * week + Condition(week) + Condition(SamE))
anova(secondaxis, strata = week, perm.max = 499, first = T)
## Permutation test for rda under reduced model
## Permutations stratified within 'week'
##
## Model: rda(formula = pyrifos ~ dose * week + Condition(week) + Condition(SamE))
## Df Var F N.Perm Pr(>F)
## RDA1 1 8.3 4.95 99 0.7
## Residual 77 129.0
He comprobado, ya dije, esto con datos propios porque carezco de los resultados del artículo de Ter Braak y van den Brink en el que están basados datos de pyrifos (solo aparecen en él las p y el porcentaje de varianza recogida por los ejes), y tanto los resultados de las F en vegan y CANOCO son similares y los eigenvalues de la prc y de la posterior rda coinciden.
Solo queda saber qué pasa con by=“axis”, porque sería bastante más cómodo hacerlo así.
Por último, como habréis comprobado el formato de las operaciones en R de este post se ve algo más vistoso. Lo he conseguido utilzando el paquete knitr y la interface gráfica RStudio. knitr le permite a uno elaborar mediante markdown informes en HTML a partir de operaciones con R, como véis, resaltando de forma diferente las órdenes en la línea de comandos y los resultados. Permite además incrustar las gráficas en el mismo documento, pero no lo tengo aún muy dominado. Por el momento, parece que el código HTML que genera es aceptado bastante bien por Blogger
jueves, 15 de noviembre de 2012
R: Curvas de Respuesta Principal (PRC) 2. Una gráfica elegante
En el pasado post hablábamos de cómo conseguir que los resultados de la función prc del paquete vegan se parecieran lo más posible a los obtenidos con CANOCO. A pesar de ello la gráfica que se obtiene directamente con la función plot queda bastante fea. Aparte del tema de colores, tamaños de fuentes y ausencia de puntos, el principal problema es la diferencia de rangos de los valores de las puntuaciones de las parcelas (observaciones, puntos de muestreo,etc.) y de las especies. De hecho, Lepš y Šmilauer utilizan un método gráfico en CANOCO que separa los ejes de ambos, de forma que pueden así tener dos escalas distintas, sin importar más que luego coincidan los dos ceros en la gráfica general.
Esta es la misma solución que propongo para obtener una gráfica más elegante y más sencilla de interpretar visualmente. Lo podemos conseguir haciendo la gráfica con plot por un lado sin dibujar las especies con el parámetro species, y haciendo una gráfica independiente para estas con linestack. Ambas gráficas tendremos que fusionarlas luego con alguna aplicación externa. Vamos a ello.
Lo primero es dejar la parte de las parcelas visualmente aceptable. Para ello necesitaremos establecer varias parámetros gráficos previos con par, después dibujarla sin la leyenda con plot y, por último, añadirle la leyenda con legend en la posición más adecuada.
library (vegan)
##Extraemos los parámetros gráficos por defecto para no perderlos
opar <- par (no.readonly=T)
##Establecemos los parámetros gráficos para que nos haga todo en negro,
##aumente un poco el tamaño de las fuentes y las líneas,suprima los
##márgenes exteriores,nos ponga márgenes adecuados para los títulos
##de los ejes y los números de las escalas, nos deje los números de
##las escalas a 0.5 y en el sentido de lectura y establecemos el
##tamaño de las marcas de escala
png ('PRC.sites2.png')
par (bty='l', col='black', cex=2, cex.axis=1, cex.lab=1.3, lwd=1.8,
xaxs='i',yaxs='i', mar=c(3,3,1,4), mgp=c(2,0.5,0), omi=c(0,0,0,0),
adj=0.5, las=1, tcl= -0.3)
##Dibujamos el gráfico sin especies y sin leyenda, con líneas y puntos y
##con límites de escala adecuados
plot (mod, scaling=1, species=F, col='black',type='b',ylab=c('PRC'),
pch=c(15,16,17,18,19), ylim=c(-1,1.8),xlim=c(-5,25),legpos=NA)
##Dibujamos la leyenda sin el nivel de control, cuidando que los
##caracteres de los puntos coincidan con los graficados
legend (0.05,0.2,legend=levels(dose)[-1],pch=c(15,16,17,18,19),
ncol=2,cex=1.5)
dev.off()
Ahora toca dibujar el eje de las especies. Si recordáis, en el post anterior extrajimos del análisis las puntuaciones de las especies a un vector con el escalado adecuado. Por si acaso, aquí está otra vez, solo que sin multiplicar por la constante correspondiente a CANOCO:
##Extraemos las puntuaciones de las especies para el primer eje
##RDA (choices=1), multiplicadas por la constante (const) que
##utiliza CANOCO
scores.sps <- scores (mod, choices=1, scaling=1,
dis='sp')
Vamos a hacer la gráfica con un poco menos de especies para que no salga tan abigarrada, así que estableceremos con colSums un límite inferior de 250, en vez de 100 como en el ejemplo de la función prc. Vamos a dibujar el eje de las especies con la función linestack, a la cual hay que especificarle que nos tiene que dibujar el eje con el parámetro axis. Con los parámetros air y hoff vamos a hacer que los nombres de las especies se separen un poco entre ellos y del eje. Por último, con el parámetro at colocaremos el eje en el centro de la gráfica. Podéis poner en el vector names los nombres completos de las especies, pero eso ya os lo dejo a vosotros.
##Dibujo del eje de especies
png ('PRCsps.png', 240, 480, res=100)
linestack (scores.sps[logabu>250], labels=names[logabu>250], axis=T,
air=1.3, hoff=6, at=-1, font=3)
dev.off()
Y ya solo tenemos que unir los dos gráficos en uno con algún programa externo. La función linestack tiene un parámetro, add, con el que añadir el eje al gráfico actual, pero si hacemos esto, por la diferencia de escalas, no nos quedará bien colocado respecto al 0 de la figura de las observaciones. Por eso es mejor hacer los dos gráficos independientemente y luego juntarlos con un programa externo. Yo lo he hecho con Libre Office Draw.
Y, en principio, ya está. Sugerencias y comentarios serán bienvenidos.